Graphbasierte Analyse von Spike-Proteinen des SARS-CoV-2
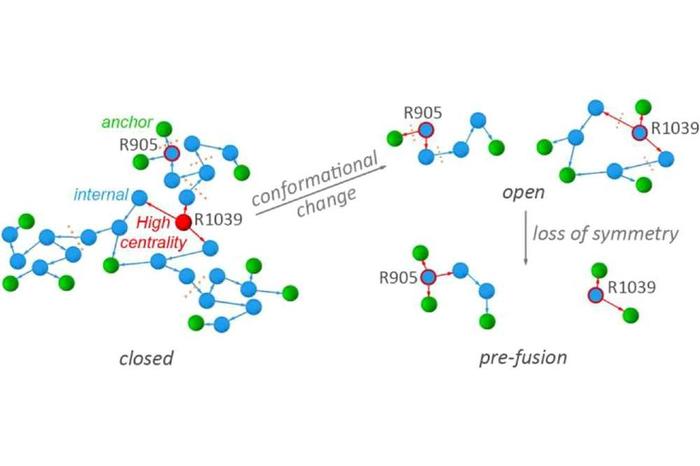
Das Forschungsteam von Professor Bondar hat einen graphbasierten Ansatz konzipiert, entwickelt und angewandt, um die strukturelle Dynamik des Spike-Proteins des SARS-CoV-2-Viruspartikels in Bezug auf die Wasserstoffbrückenbindungen zu untersuchen. Das Team identifizierte ein komplexes, ausgedehntes Netzwerk von Wasserstoffbrückenbindungen an der Bindungsschnittstelle zwischen einem Spike-Protein und einem ACE2-Rezeptor.
News vom 10.12.2020
Die Oberfläche des SARS-CoV-2-Viruspartikels ist mit den sogenannten Spike-Proteinen, oder S-Proteinen, ausgestattet, die an die ACE2-Rezeptoren der Wirtszelle andocken. Der Mechanismus, der bestimmte Anordnungen eines S-Proteins für die Bindung an den Rezeptor auswählt, könnte die Entwicklung von Peptiden zur Verhinderung der Bindung voranbringen.
Computergestützte Ansätze ermöglichen es, ein detailliertes molekulares Bild der Interaktionen, die die Bindung von S-Protein an den Wirtsrezeptor steuern, zu liefern. Die enorme Größe des S-Proteins macht es jedoch schwierig, die relative Bedeutung bestimmter Regionen oder Gruppen des Proteins zu bewerten. Um dieses Problem zu lösen, arbeitete das Forschungsteam von Professor Ana-Nicoleta Bondar mit einer graphbasierten Methode. Das Team von Doktoranden Konstantina Karathanou, Michalis Lazaratos, Krzysztof Buzar sowie Studierenden Eva Bertalan und Malte Siemers analysierte mit dem Ansatz die strukturelle Dynamik des S-Proteins in Bezug auf die Wasserstoffbrückenbindungen.
Das Forschungsteam fand heraus, dass der Übergang des S-Proteins von einer geschlossenen Konformation zu einer Präfusionskonformation, welche einen S-Protein binden kann, mit dem Verlust der dreifachen Kompositionssymmetrie von Wasserstoffbrückenbindungsclustern an den Schlüsselstellen des S-Proteins einhergeht. Ein komplexes, ausgedehntes Netzwerk von Wasserstoffbrückenbindungen wurde an der Bindungsschnittstelle zwischen Spike-Protein und ACE2 identifiziert. In diesem Netzwerk nimmt der Aminosäurerest des S-Proteins N501 eine zentrale Rolle ein. Interessanterweise gehört N501 zu den drei Gruppen, die in der neuen 501Y.V2 SARS-CoV-2 Linie mutiert sind (Tegally et al, medRxiv, https://doi.org.10.1101/2020).
Die Forschungsergebnisse wurden in der Fachzeitschrift Journal of Structural Biology veröffentlicht.
Schlagwörter
- Biophysik
- Coronavirus
- Grundlagenforschung
- Proteine
- SARS-CoV-2
- Spike-Protein